今日《自然》发布迄今最大规模人类遗传变异体数据库
今日,顶尖学术期刊《自然》的最新一期如约上线,登上封面的主题是“基因组聚集数据库(gnomAD)”。这份由多国研究者联合发展起来的公开目录,是迄今为止最大规模的人体遗传变异体数据库,汇集了超过14万人的基因组测序或外显子组测序数据。

在本期《自然》杂志上,一共发表了4篇相关论文。此外,在Nature Medicine、Nature Communications等子刊上也有同系列的论文。在今天的这篇文章中,学术经纬团队将和读者朋友们一起来了解堪称人类遗传学研究里程碑的gnomAD数据库。

基因测序技术的出现让我们可以读出一个人所有的遗传信息——人类基因组。不过,相比测出基因序列,更大的挑战是了解这些基因的生理功能。对于人类基因组中大多数基因的功能,我们依然知之甚少。

揭示基因功能的一种方法是观察基因突变后的结果。这些基因变异体,常常会让其编码产生的蛋白质失活,因此被称为功能丧失型(loss-of-function, LoF)变异体。但这类变异体在人群中较为罕见,这意味着,想要发现变异体,评估考察每一种变异体带来的结果,就需要非常大的基因组样本量。这也正是大规模数据库的意义所在。

基因组聚集数据库(gnomAD)项目,通过各种大型人群测序项目汇集数据,来鉴定各种功能丧失型变异体。
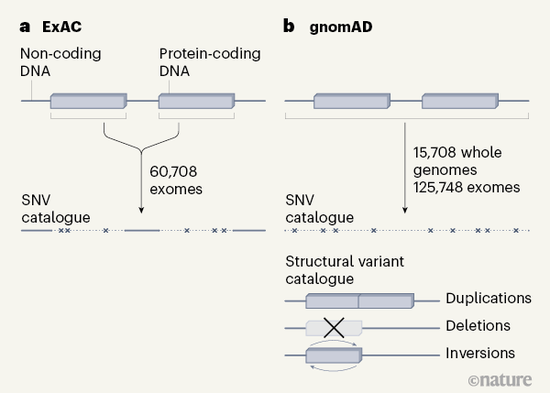
gnomAD项目之前,科学家们在2016年公开了外显子组聚集联盟(ExAC),收录有6万多个外显子组数据,主要是基因组中与蛋白质合成直接相关的DNA片段(外显子)。根据《自然》的一篇概述性文章的介绍,新的gnomAD不仅汇集了125748个全外显子组序列,还包含了15708个全基因组测序数据,规模和范围都有增加,因此可以系统性地记录更多样、更复杂的基因变异体,并了解蛋白质编码序列以外的变异。

▲相比ExAC,gnomAD的规模和范围更大,可以解读的基因变异体也更丰富(图片来源:参考资料[5])
研究团队从中总共筛选出了443769个预测的功能丧失型(predicted LoF, pLoF)变异体,预测这些变异体会影响其编码蛋白的正常运作。研究人员进而对这些变异体进行了分类,从对生理机能几乎没影响到导致严重的健康问题,以便更好地发现造成常见遗传病和罕见遗传病的基因。


同系列的第三篇论文探讨了如何利用人类功能丧失型变异体数据库识别候选药物靶点。研究人员报告了几个关键发现:首先,那些不能容忍有功能丧失型变异体的基因(也就是必需基因),依然可以作为可行的成功靶点。去设计抑制剂的开发。具体来看,当发现某些个体在特定基因上携带了两个pLoF变异体时,这个基因可能会是一个好的药物靶点。;其次,大部分基因中功能丧失型变异体十分罕见,研究人表明在推断这类变异体时会有很多误判,因此要收集到确切证据,需要比gnomAD样本量大1000倍的队列来进行验证;第三,我们虽然可以对变异体进行自动的标注,但为了去除人为引入的误差,我们依旧需要人工对其进行检查。
来源:学术经纬
《自然》同时发表的第四篇论文中,研究人员们分析了gnomAD数据库中的近1.5万个全基因组测序数据,创建了一个结构变异资源库。
结构变异(SV)指的是染色体上有大片段DNA发生了重新排列,有缺失、重复、插入、易位甚至是颠倒方向等多种类型。这类变异是很多遗传病和癌症的重要诱因。研究人员指出,这个包含43.3万种SV的丰富资源库,“在人群遗传学,疾病关联研究和诊断筛查中具有广泛的用途”。
《自然》同期的一篇评论文章指出,这一大规模的基因组测序和分析工作产生了迄今为止最全面的数据和工具来了解人类的遗传变异。gnomeAD已经将这些数据和工具公开。这份宝贵的遗传资源,将改变我们解读个体基因组的方式,为我们理解人类的生物学特征和疾病,评估罕见和常见遗传病,提供重要信息。
来源:学术经纬

 微信公众号
微信公众号 京公网安备11010502033649
京公网安备11010502033649